CarrierSeq™

Для скрининга носительства генетических заболеваний с помощью полупроводникового секвенирования
Предназначен для расширенного скрининга на широкий спектр наследственных заболеваний. Исследование выполняется без учета расовых или этнических особенностей. Позволяет стандартизировать и расширить применяемые в лабораториях решения для выявления носителей наследственных заболеваний.
Целью скрининга носителей является выявление пар, находящихся в группе риска передачи серьезных генетических заболеваний, таких как тяжелые моногенные нарушения с аутосомно-рецессивным (например, кистозный фиброз) или рецессивным, связанным с Х-хромосомой (например, мышечная дистрофия Дюшенна), типами наследования.
Тщательно разработанный продукт для точного выявления носителей
В основе набора лежит использование технологии таргетной (целевой) амплификации участков генома человека и последующего создания таргетной ДНК-библиотеки (технология AmpliSeq). Для этого используется панель праймеров Ion AmpliSeq CarrierSeq ECS Panel.
Панель Ion AmpliSeq CarrierSeq ECS Panel
- Обеспечивает исчерпывающий охват распространенных и редких вариантов, помогая достичь более высокого уровня выявления нарушений.
- Нацелена на более чем 14 000 ампликонов, которые покрывают все кодирующие области 420 целевых генов, включая границы интронов/экзонов, для генотипирования от 28 000 SNV и инделов из архива ClinVar.
- Обеспечивает надежное нацеливание для анализа CNV для максимального обнаружения статуса нoсителей.
Наличие некоторых генетических вариантов, связанных с наиболее серьезными заболеваниями, сложно установить только при помощи методов секвенирования нового поколения (NGS), поэтому часто требуются автономные дополнительные анализы. Панель Ion AmpliSeq CarrierSeq объединяет эти автономные анализы в единый, включая такие гены, как:
- SMN1 для спинальной мышечной атрофии,
- GBA для болезни Гоше,
- CYP21A2 для врожденной гиперплазии надпочечников с дефицитом 21-гидроксилазы,
- HBA1 и HBA2 для альфа-талассемии.
Анализ технически сложных генов
В CarrierSeq используется комбинация чтения последовательности и анализ количества копий на основе глубины, однонуклеотидное генотипирование и специализированные алгоритмы определения вариантов в трех классах «особых» генов.
SMN1 (Спинальная мышечная атрофия)
Целевое количество копий и анализ генотипирования SNV используются для определения количества копий экзона 7 с помощью специального анализа количества копий (CN) для SMN1 и SMN2.
HBA1 и HBA2 (Альфа-талассемия)
Небольшие гены, отличающиеся только в экзоне 3. Специальный алгоритмический анализ определяет количество копий в этой гомологичной области.
Гены с высокой степенью гомологии GBA и CYP21A2 (21-OH CAH)
Анализ в первую очередь рассматривает количество копий гена и возможные события конверсии гена как механизмы мутации.
Состав набора
Набор содержит все необходимые компоненты для создания ДНК-библиотеки, подготовки темплейта для секвенирования с помощью автоматизированной станции и последующего секвенирования. Предлагаются версии набора с 530 или 540 чипом, позволяющие за один запуск исследовать от 6 до 32 образцов (по два чипа за один запуск).
+ чип 530
+ чип 540
Анализ данных
Локальные и облачные программные инструменты позволяют максимально ускорить и упростить анализ полученных данных секвенирования. Доступ к программам осуществляется по локальной сети с рабочих мест в соответствии с предоставленными правами.
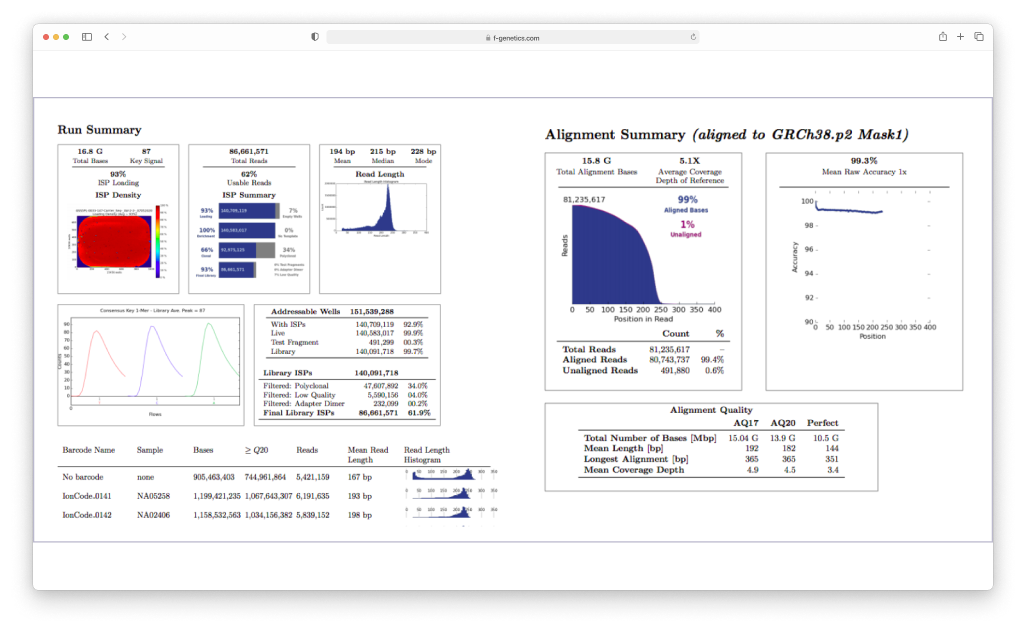
ПО Torrent Suite систем F-Genetics установлено на встроенный компьютер генетического секвенатора
ПО предназначено для планирования запусков, мониторинга, протоколирования и анализа качества секвенирования
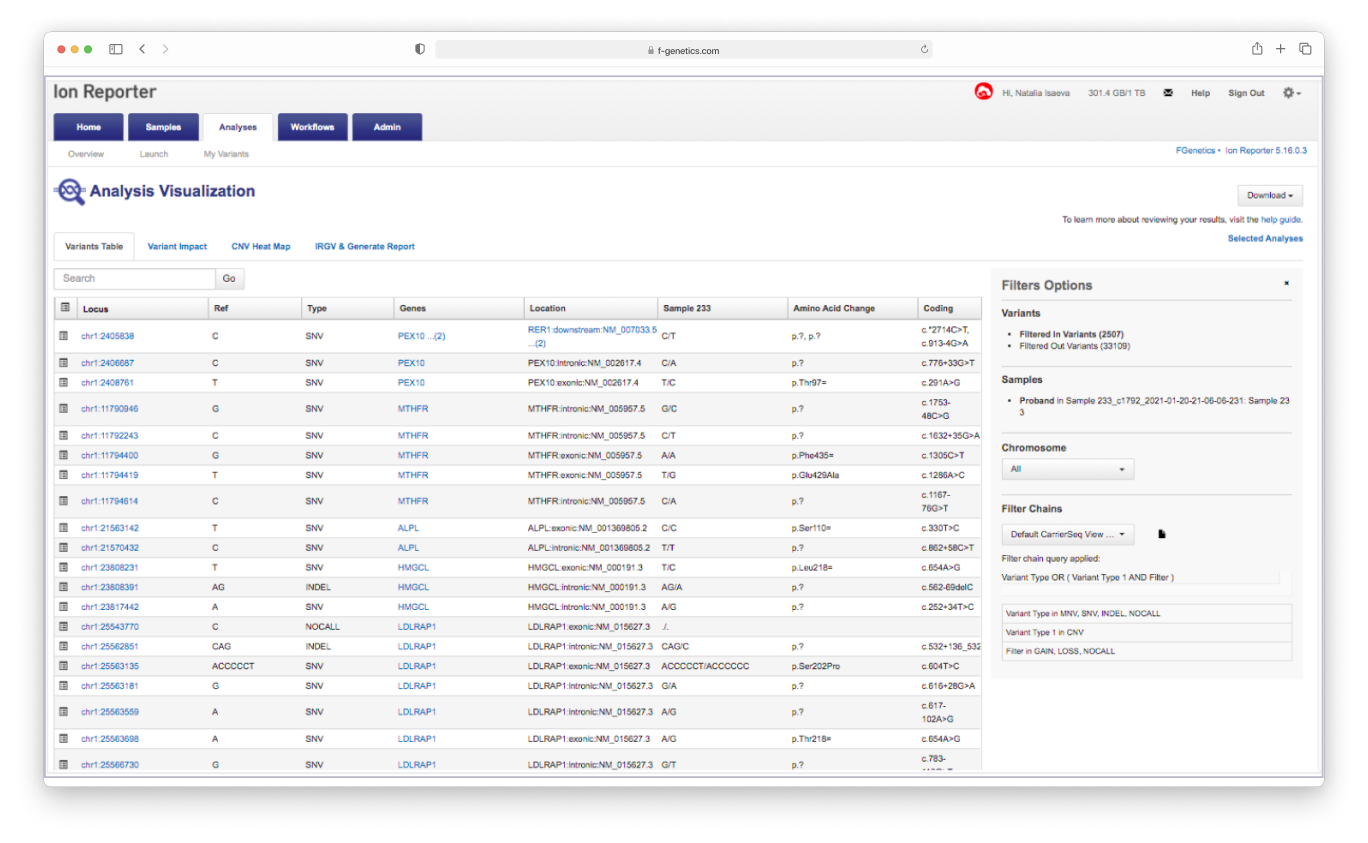
Полный перечень найденных вариантов по каждому образцу выдается в виде таблицы
Ion Reporter Server System автоматически распознает различные типы мутаций с помощью специально настроенного процесса обработки данных.
Система устанавливается на локальный сервер или доступна бесплатно в формате облачного решения.
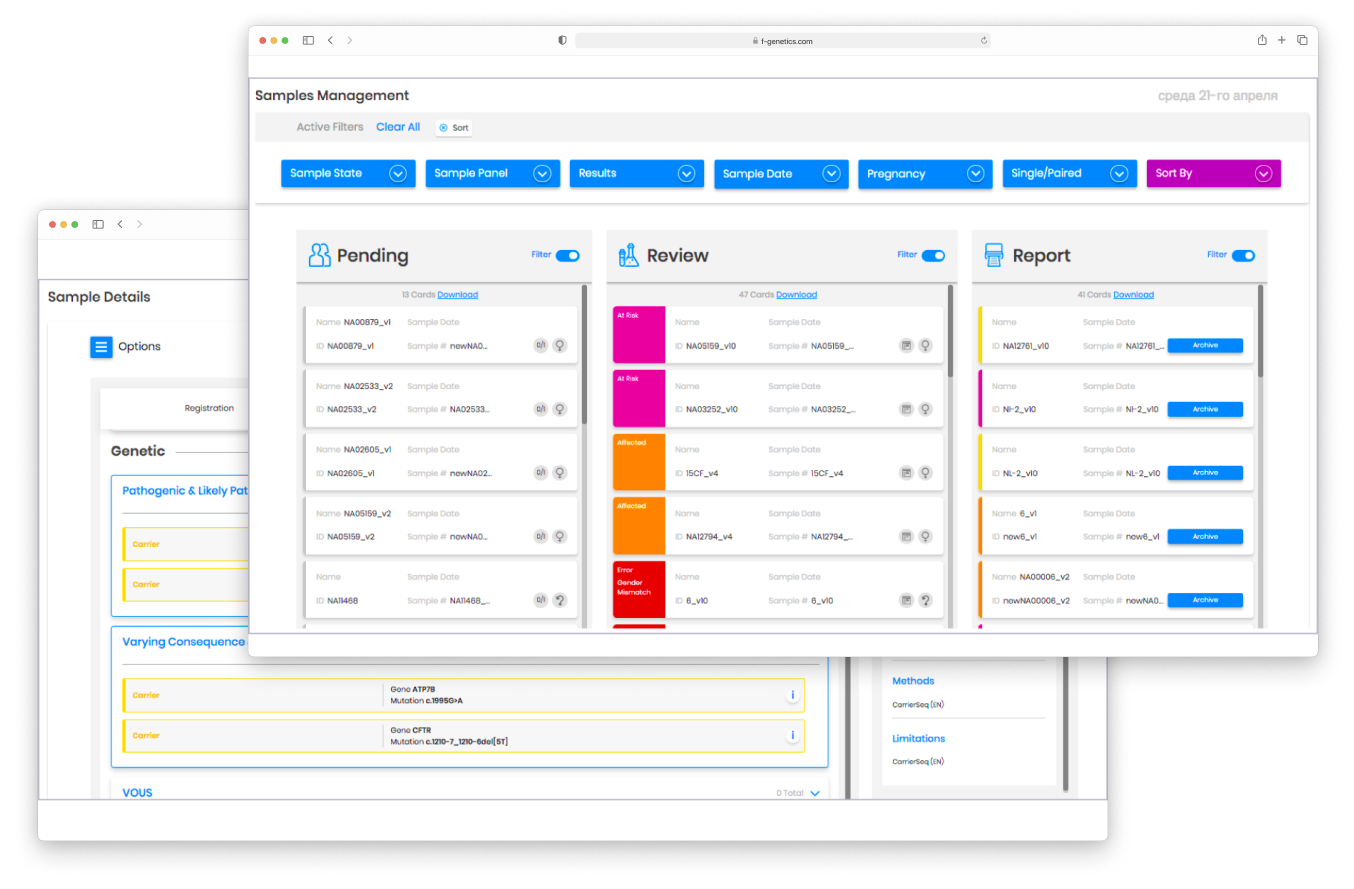
Для итоговой обработки данных и формирования отчета используется программное обеспечение Carrier Reporter
Интерфейс позволяет анализировать результаты скрининга носительства и составлять отчеты
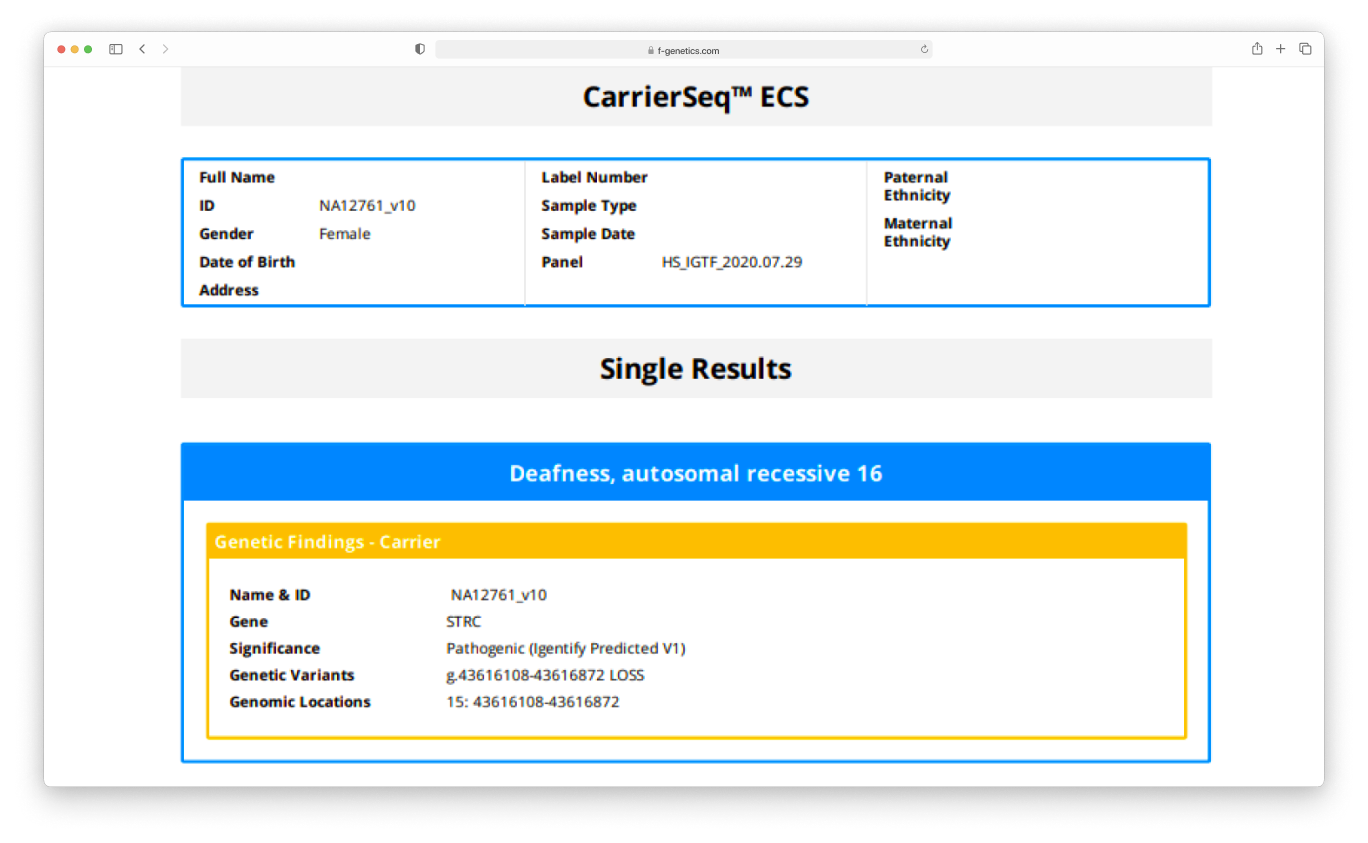
Специальные алгоритмы направлены на выявление различных типов мутаций CNV, SNV/indels в технически сложных для анализа генах, таких как: CYP21A2, GBA, HBA1/2 и SMN1
Полноценный набор инструментов для быстрого перевода найденных вариантов в аннотированный отчет.
Облачная или локальная версия
Два поддерживаемых варианта выявления генетических вариантов:
- целевое выявление по горячим точкам (включает возможность настройки параметров),
- поиск неизвестных вариантов.
Анализ двугенных вариантов (одновременные мутации в двух генах, способствующие развитию заболевания), таких как GJB2 и GJB6; HBA1 и HBA2.